논문 정보
- Title: Predicting transcriptional responses to novel chemical perturbations using deep generative model for drug discovery
- Authors: Xiaoning Qi, Lianhe Zhao, Chenyu Tian, Yueyue Li, Zhen-Lin Chen, Peipei Huo, Runsheng Chen, Xiaodong Liu, Baoping Wan, Shengyong Yang, Yi Zhao
- Journal: Nature Communications, Volume 15, Article 9256 (2024)
- DOI: 10.1038/s41467-024-53457-1
연구 배경 및 동기
신약 개발에서 화학 물질이 세포의 전사체(transcriptome)에 미치는 영향을 이해하는 것은 핵심적인 과제다. 그러나 모든 질병-화합물 조합에 대해 실험적으로 스크리닝하는 것은 현실적으로 불가능하다. 예를 들어, L1000 프로젝트는 82개 세포주에서 175,549개의 생물활성 화합물에 대한 883,269개의 전사체 프로파일을 생성했지만, 이는 전체 가능한 조합의 극히 일부에 불과하다.
이러한 한계를 극복하기 위해, 저자들은 PRnet (Perturbation Response Network)을 제안한다. 이는 perturbation-conditioned deep generative model로, 실험적으로 한 번도 테스트되지 않은 새로운 화합물에 대한 전사체 반응을 bulk 및 single-cell 수준에서 예측할 수 있다.
모델 아키텍처
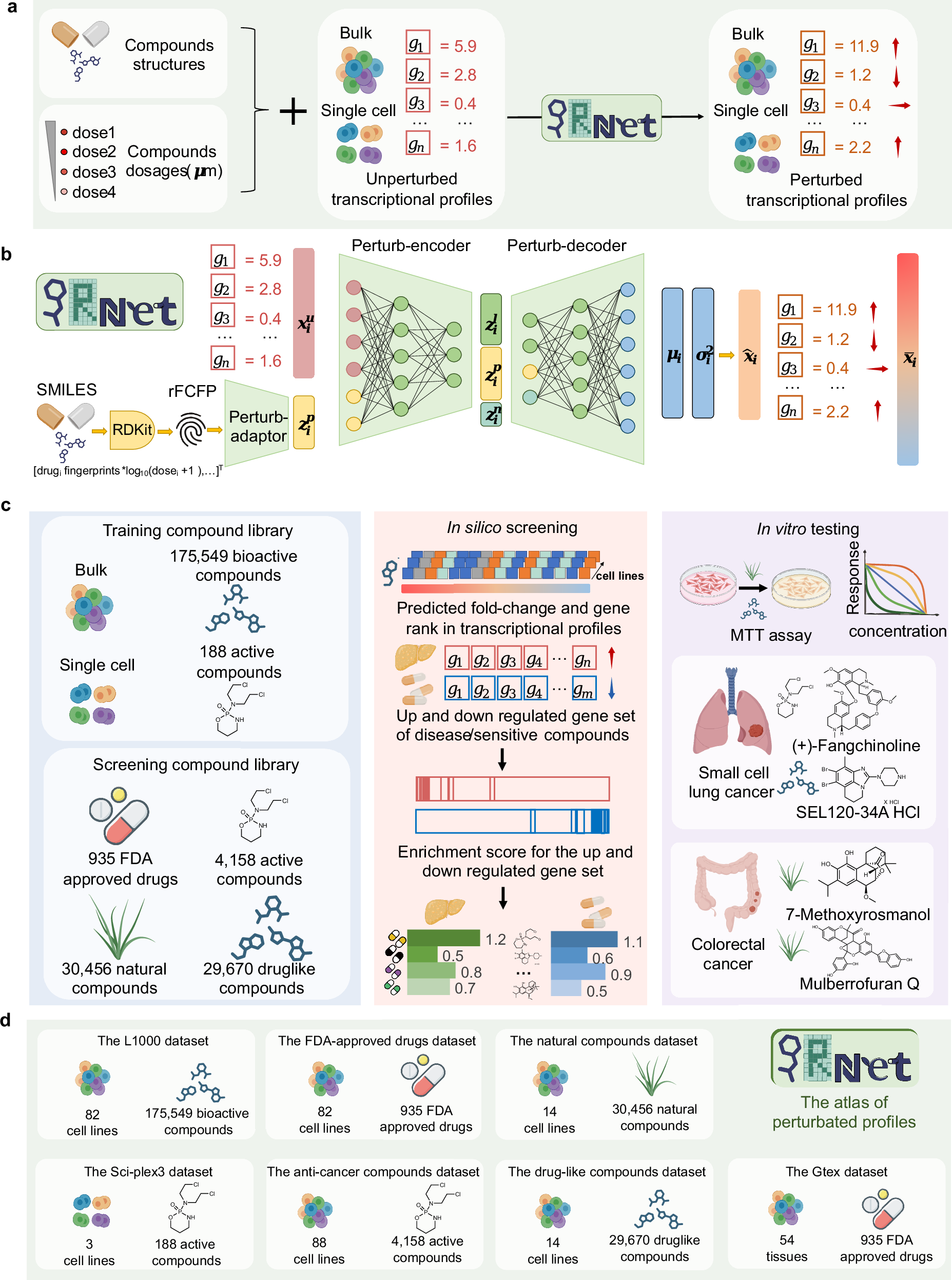 Figure 1: PRnet의 전체 아키텍처와 워크플로우 개요 (출처: Qi et al., Nat Commun 2024)
Figure 1: PRnet의 전체 아키텍처와 워크플로우 개요 (출처: Qi et al., Nat Commun 2024)
PRnet은 Variational Autoencoder (VAE) 기반의 생성 모델로, 세 가지 핵심 모듈로 구성된다:
1. Perturb-adapter
- 화합물의 구조 정보(molecular fingerprint)와 농도(dosage) 정보를 인코딩
- 화합물의 로그 농도를 약물 feature의 가중치로 사용하여 dosage-dependent한 반응을 모델링
2. Perturb-encoder
- 비섭동(unperturbed) 전사체 프로파일과 섭동 조건을 입력으로 받아 잠재 공간(latent space)으로 매핑
- Variational inference를 통해 전사체 반응의 분포를 학습
3. Perturb-decoder
- 잠재 공간의 표현과 섭동 조건으로부터 섭동 후의 전사체 프로파일을 생성
- Gene-level의 반응 해석이 가능
이 구조의 핵심은 data-driven approach로, 화합물 구조와 전사체 데이터의 관계를 학습하여 완전히 새로운 화합물에 대해서도 일반화(generalization)할 수 있다는 점이다.
학습 데이터
PRnet은 두 가지 대규모 데이터셋으로 학습되었다:
| 데이터셋 | 수준 | 프로파일 수 | 세포주 | 화합물 수 |
|---|---|---|---|---|
| L1000 | Bulk | 883,269 | 82 | 175,549 |
| Sci-Plex3 | Single-cell | 290,888 | 3 | 188 |
주요 결과
1. 기존 방법 대비 우수한 예측 성능
PRnet은 새로운 화합물, 새로운 경로(pathway), 새로운 세포주에 대한 전사체 반응 예측에서 기존 방법들(CPA, chemCPA, TranSiGen 등)을 유의미하게 능가했다. 특히, unseen cell line에 대한 log(FC) 예측에서 PCC(Pearson Correlation Coefficient)가 0.3 이상 향상되었다.
2. Gene-level 반응 해석
PRnet은 단순 예측을 넘어 유전자 수준에서의 반응 해석을 가능하게 한다. 특정 화합물이 어떤 유전자의 발현을 상향/하향 조절하는지 예측하고, 이를 기반으로 작용 기전(mechanism of action)을 추론할 수 있다.
3. In-silico 약물 스크리닝
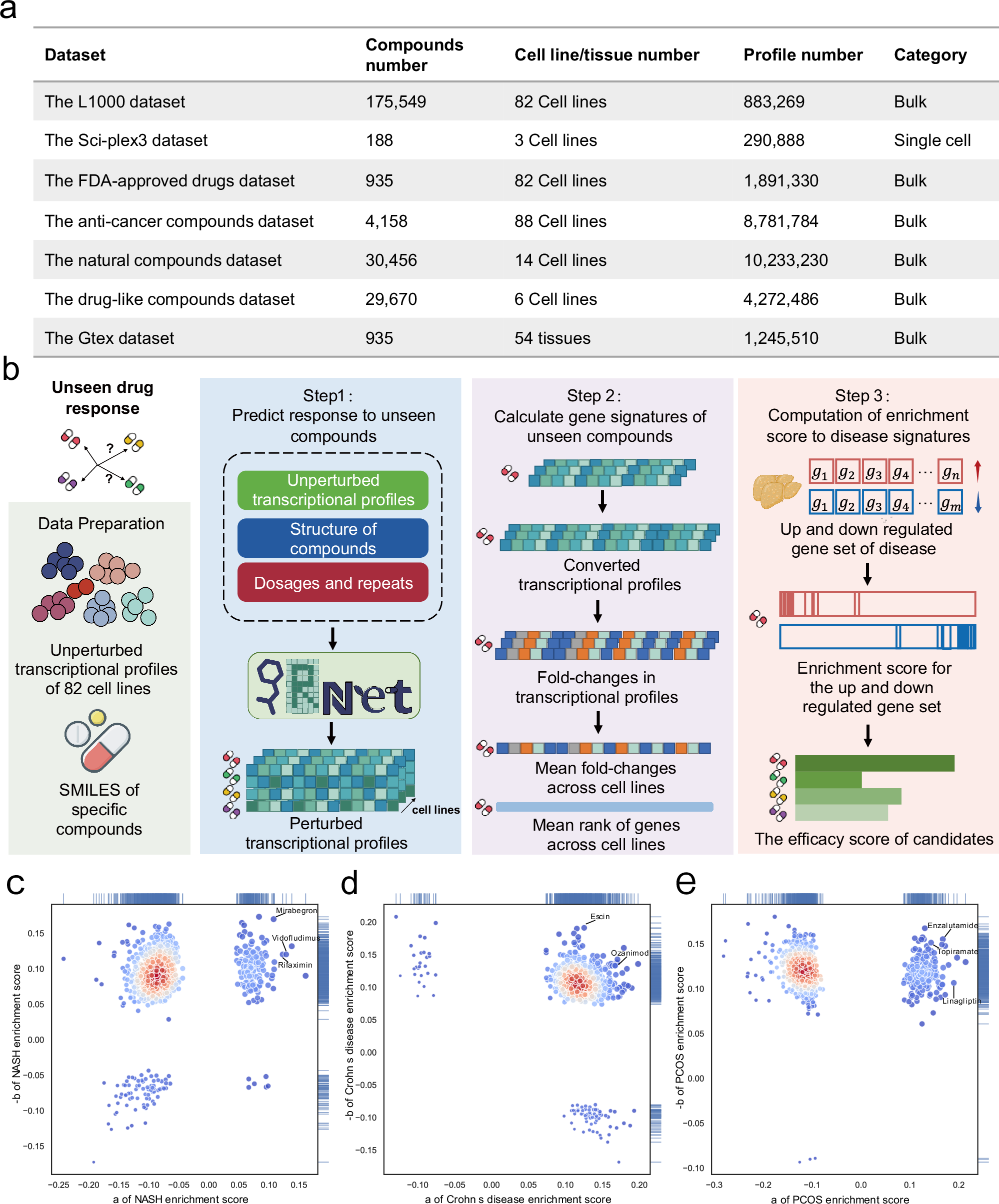 Figure 5: PRnet 기반 약물 후보 추천 및 실험적 검증 (출처: Qi et al., Nat Commun 2024)
Figure 5: PRnet 기반 약물 후보 추천 및 실험적 검증 (출처: Qi et al., Nat Commun 2024)
PRnet의 in-silico 스크리닝 워크플로우는 다음과 같다:
- Step 1: 스크리닝 라이브러리의 화합물 구조를 입력으로, 82개 세포주에서 다양한 농도 구배에 대한 전사체 프로파일 예측
- Step 2: 특정 질병의 gene signature를 기반으로, 상향/하향 조절 유전자 세트에 대한 enrichment score 계산
- Step 3: Enrichment score 기반으로 화합물 순위 결정
4. 실험적 검증 — 암 치료 후보 물질 발굴
PRnet이 추천한 화합물 후보에 대해 in vitro MTT assay를 통한 실험적 검증을 수행했다:
- 소세포폐암 (SCLC): (+)-Fangchinoline과 SEL120-34A HCl이 유의미한 항암 활성 확인
- 대장암 (CRC): 7-Methoxyrosmanol과 Mulberrofuran Q의 항암 활성 확인
이는 PRnet의 in-silico 예측이 실제 실험 결과와 일치함을 보여주는 중요한 검증이다.
5. 대규모 Perturbation Atlas 구축
PRnet을 활용하여 88개 세포주, 52개 조직을 포괄하는 대규모 perturbation profile atlas를 생성했다. 이 atlas는 다음을 포함한다:
- L1000 및 Sci-Plex3 데이터셋
- FDA 승인 약물 라이브러리
- 항암 화합물 라이브러리
- 천연물 라이브러리
- Drug-like 화합물 라이브러리
- GTEx 조직 발현 데이터
최종적으로, PRnet은 233개 질병에 대한 약물 후보를 추천할 수 있는 워크플로우를 제공한다.
의의 및 한계
의의
- 확장성: 수십만 개의 화합물에 대한 전사체 반응을 computational하게 예측 가능
- 일반화 능력: 학습 데이터에 없는 새로운 화합물, 새로운 세포주에 대해서도 예측 가능
- 실험적 검증: In-silico 예측을 실제 실험으로 검증하여 모델의 실용성 입증
- 다중 스케일: Bulk과 single-cell 수준 모두에서 작동
한계 및 후속 연구 방향
- Over-denoising 문제: 최근 Nature Machine Intelligence에 발표된 XPert 논문에서 지적된 바와 같이, VAE 기반 모델은 과도한 denoising으로 인해 중요한 생물학적 정보가 손실될 수 있음
- 세포 맥락 의존성: 동일 화합물이라도 세포 유형에 따라 반응이 크게 달라질 수 있어, 더 다양한 세포 맥락에 대한 학습 필요
- In vivo 검증: 현재 in vitro 수준의 검증에 머물러 있어, 동물 모델 및 임상 데이터와의 연결이 필요
개인적 소감
이 논문은 전사체 수준의 약물 반응 예측이라는 challenging한 문제에 대해 생성 모델 기반의 체계적인 솔루션을 제시한다. 특히 인상적인 부분은:
- L1000이라는 대규모 데이터셋을 효과적으로 활용한 점
- 단순 예측을 넘어 233개 질병에 대한 약물 추천 파이프라인까지 구축한 점
- 실험적 검증을 통해 computational prediction의 실용성을 보여준 점
Aging research와 bioinformatics 관점에서, PRnet과 같은 모델을 노화 관련 질환의 약물 스크리닝에 적용하면 anti-aging 약물 후보 발굴을 가속화할 수 있을 것으로 보인다. 특히 노화 관련 gene signature를 활용한 enrichment score 기반 스크리닝은 바로 적용 가능한 전략이다.
References
- Qi, X., Zhao, L., Tian, C. et al. Predicting transcriptional responses to novel chemical perturbations using deep generative model for drug discovery. Nat Commun 15, 9256 (2024). https://doi.org/10.1038/s41467-024-53457-1
-
PubMed PMC Full Text
💬 댓글